2021年8月12日,清华大学药学院胡泽平课题组与合作者在《Nature Metabolism》杂志以研究长文(Article)在线发表题为“Aberrant NAD+ metabolism underlies Zika virus-induced microcephaly” 的文章,揭示ZIKV感染导致小头症脑组织发生显著的烟酰胺腺嘌呤二核苷酸(NAD+)代谢重编程;并发现靶向干预NAD+代谢可以在动物模型中有效改善ZIKV感染所致的小头症。

孕期感染寨卡病毒(Zika virus,ZIKV)会导致新生儿小头畸形[1, 2]。然而,其潜在的发生机制尚不明确,也缺乏有效的治疗策略。病毒感染宿主后,可以通过“劫持”并重编程宿主的代谢以满足其快速生长复制的物质和能量需求,并促使其发生免疫逃逸[3, 4],而靶向代谢可以有效抑制病毒复制[5-8]。因此,探究ZIKV感染对宿主代谢的重编程和影响,有助于理解小头症的发生机制,进而开发潜在的治疗策略。近年来,多组学整合研究策略在理解复杂疾病(如肿瘤[9],糖尿病[10],病毒感染等[11, 12])发生发展的分子机制中发挥着越来越重要的作用,也为发现疾病治疗的潜在靶标提供了重要工具和理论基础。
为了探究ZIKV感染所致小头症的分子机制和潜在治疗靶点,作者利用包括转录组学,蛋白质组学,磷酸化蛋白质组学,以及代谢组学在内的多组学技术,全面描绘了ZIKV感染脑组织中的分子变化特征。转录组学和蛋白组学揭示了ZIKV感染脑组织引起了显著的细胞凋亡和免疫反应。蛋白组学揭示了ZIKV感染脑组织中胆固醇代谢上调和脑神经发育障碍。进一步,通过将代谢组学与其他组学在通路水平进行多组学整合分析,作者从不同分子层面上交互验证了ZIKV感染导致小头症的发生过程中的一系列代谢重编程事件,如NAD+代谢,三羧酸循环(TCA cycle),氧化磷酸化(OXPHOS),嘌呤和嘧啶代谢等,同时也提示ZIKV感染可能导致线粒体功能发生障碍(图1)。值得注意的是,ZIKV感染脑组织中NAD+水平急剧下降,而从头合成和补救合成途径上的前体(tryptophan、kynurenine、nicotinamide riboside、nicotinamide mononucleotide)水平均发生不同程度的上调(图1)。因此推测NAD+代谢通路重编程可能是ZIKV感染所致小头症的关键节点和潜在干预靶标。此外,磷酸化蛋白质组学揭示了ZIKV感染脑组织中MAPK通路显著激活,结合NAD+代谢以及相关蛋白变化,推测ZIKV感染可能存在MAPK-NMNAT2-NAD+轴的潜在分子机制。

图1 ZIKV感染引起脑内NAD+代谢,TCA cycle以及OXPHOS代谢重编程
为了探究NAD+代谢重编程在ZIKV感染所致小头症中的功能,作者使用ZIKV感染小鼠所致小头症模型进行体内实验,观察回补NAD+及相关前体是否可以改善ZIKV感染所致的小头症。研究结果表明,脑注射NAD+可以显著抑制ZIKV感染所致的细胞凋亡,增加大脑皮层厚度。而腹腔注射NAD+的重要前体nicotinamide riboside(NR)不仅可以显著抑制ZIKV感染所致的细胞凋亡,增加大脑皮层厚度,还可以增加感染小鼠的体重和脑重,延长其生存期(图2)。
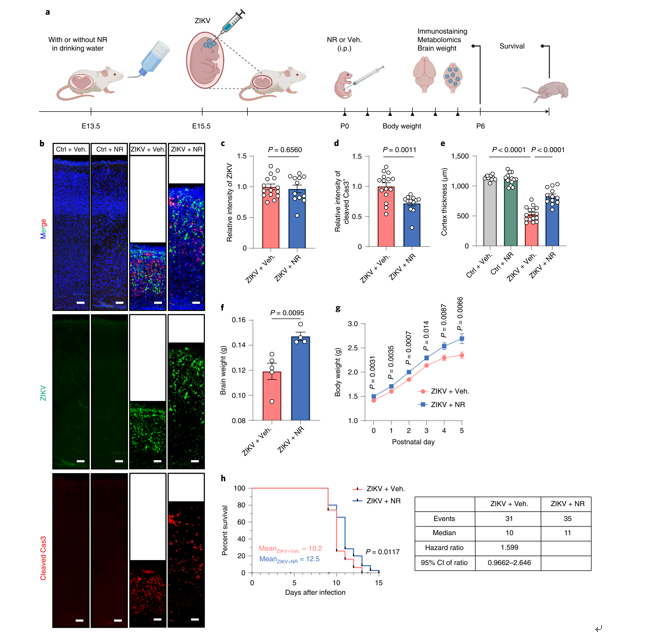
图2 NR可以显著改善ZIKV感染导致的小头症
综上,该研究通过对ZIKV感染所致小头症的多组学分析,揭示了ZIKV感染导致显著的NAD+代谢重编程,并证明干预NAD+代谢能够从分子和整体层面上显著改善ZIKV感染所致的小头症。该研究不仅提供了ZIKV感染后不同分子层面重塑的组学大数据信息,也为理解ZIKV感染所致小头症的分子机制提供了新见解,并提示干预NAD+代谢或可作为治疗ZIKV感染所致小头症的潜在新策略。
清华大学药学院博士后庞欢欢、聂萌、博士生李杰,中国科学院遗传与发育生物学研究所博士生姜义圣,清华大学生命科学学院博士生王钰珅为该论文的共同第一作者。清华大学药学院胡泽平研究员、中国科学院遗传与发育生物学研究所许执恒研究员、完美体育·(中国)官方网站蓝勋研究员、国家蛋白质中心宋雷博士为该论文的共同通讯作者。清华大学李蓬教授和李梢教授参与合作并给予指导和协助。清华大学施一公教授、曾文文教授、王戈林教授等对该研究提供了大力支持和帮助。该研究工作得到了科技部“发育编程及其代谢调节”重点专项、国家自然科学基金委“糖脂代谢的时空网络调控”重大计划的重点项目、国家自然科学基金委面上项目、清华-北大生命联合中心,以及中国科学院等项目支持。
原文链接:https://www.nature.com/articles/s42255-021-00437-0
参考文献:
1.Rostaing, L.P. and P. Malvezzi, Zika Virus and Microcephaly. N Engl J Med, 2016. 374(10): p. 982-4.
2.Lessler, J., et al., Assessing the global threat from Zika virus. Science, 2016. 353(6300): p. aaf8160.
3.Olive, A.J. and C.M. Sassetti, Metabolic crosstalk between host and pathogen: sensing, adapting and competing. Nat Rev Microbiol, 2016. 14(4): p. 221-34.
4.Wang, A., H.H. Luan, and R. Medzhitov, An evolutionary perspective on immunometabolism. Science, 2019. 363(6423).
5.Kilbourne, E.D., Inhibition of influenza virus multiplication with a glucose antimetabolite (2-deoxy-D-glucose). Nature, 1959. 183(4656): p. 271-2.
6.Bojkova, D., et al., Proteomics of SARS-CoV-2-infected host cells reveals therapy targets. Nature, 2020.
7.Xiao, N., et al., Integrated cytokine and metabolite analysis reveals immunometabolic reprogramming in COVID-19 patients with therapeutic implications. Nature communications, 2021. 12(1): p. 1618.
8.Li, X.K., et al., Arginine deficiency is involved in thrombocytopenia and immunosuppression in severe fever with thrombocytopenia syndrome. Sci Transl Med, 2018. 10(459).
9.Gao, Q., et al., Integrated Proteogenomic Characterization of HBV-Related Hepatocellular Carcinoma. Cell, 2019. 179(2): p. 561-577 e22.
10.Zhou, W., et al., Longitudinal multi-omics of host-microbe dynamics in prediabetes. Nature, 2019. 569(7758): p. 663-671.
11.Eisfeld, A.J., et al., Multi-platform 'Omics Analysis of Human Ebola Virus Disease Pathogenesis. Cell Host Microbe, 2017. 22(6): p. 817-829 e8.
12.Shen, B., et al., Proteomic and Metabolomic Characterization of COVID-19 Patient Sera. Cell, 2020.
